染色体核型分析是细胞遗传学的主要技术之一,以中期染色体为研究对象,对染色体进行分析、比较、排序和编号,这对研究人类遗传病的机制、物种亲缘关系与进化、远缘杂交的鉴定等都有重要的意义.染色体通常只能在细胞分裂中期才能在光镜下看到,此时染色体呈圆柱状或杆状,以浓缩形式排列在细胞中心.图1a是光学显微镜下拍摄的染色体中期图像,图1b是医生在辅助设备的帮助下对染色体中期图像进行分割和分类形成的核型分析图片.从图1a中展示的染色体中期图像可以看到,对染色体中期图像进行人工分割和分类处理主要有以下难点:(1)染色体中期图像包含较多的染色体,分割出所有的染色体需要耗费大量的时间和精力,杂质的存在和染色体之间的接触和重叠也增加了分割工作的难度;(2)需要专业的医生花费大量的时间和精力,根据不同类别染色体的形态结构对分割后的单条染色体进行分类,而一些染色体由于玻片制作的原因导致形态结构的差异较小,使分类难度增大,分类准确度降低.
为了实现自动核型分析,减少人力成本,Sharma et al[1]提出一种基于众包的染色体分割方法,首次使用卷积神经网络(Convolutional Neural Networks,CNN)实现染色体的分类,还提出染色体伸直和长度归一化等几种预处理方法.但由于他们的方法的分割过程需要人工参与,分类精度较低,不能满足自动核型分析的要求.为了提高染色体分割和分类的准确度,减少核型分析过程中的人工参与,本文对他们的方法进行了改进,旨在提高自动核型分析的准确性和可用性.
本文提供了一种深度学习方法进行核型分析.首先将染色体中期图像输入分割网络进行实例分割,提取出每一条染色体个体;然后将提取出的染色体送入多输入的CNN进行核型分析;同时,针对弯曲的染色体个体设计了一种基于几何的优化算法,在分类处理前,算法将每条染色体进行伸直优化操作.
本文的主要贡献如下:
(1)使用Mask R⁃CNN(Region⁃Convolutional Neural Networks)[2]完成从染色体中期图像中分割出染色体的实例分割任务,效果较好.
(2)提出一种基于中轴的染色体伸直优化算法,提高了分类精度,在实际应用中保证了智能系统具有优秀的可用性.
(3)提出多输入的CNN网络来实现染色体分类的任务.通过大量实验证明该网络的性能比典型的深度神经网络相关算法有显著的提高.
1 相关工作
染色体的核型分析有两个重要步骤:染色体的分割和分类.为了减轻核型分析的工作量,研究人员提出了一些自动分割和分类的方法.
1.1 染色体分割
随着深度学习的发展,CNN在许多计算机视觉任务上取得了优异的成绩.在医疗图像处理领域,学者们也开始尝试用CNN完成医疗图像的分割.BenTaieb and Hamarneh[6]优化了FCN(Fully Convolutional Network)[7]的结构并成功将其应用于医疗图像分割领域.医学图像分割网络U⁃net[8]在Hele细胞分割和神经元结构分割任务中取得了很好的效果.上述医疗图像的分割均为语义分割,而本文是要从染色体中期图像中识别出多个染色体目标并分割出单个染色体,这属于实例分割.本文首次将深度学习应用于染色体分割领域,在选择分割网络时比较了Mask R⁃CNN[2],FCIS(Fully Convolutional Instance⁃aware Semantic Segmentation)[9]和ShapeMask[10].这三个网络均能实现实例分割,但ShapeMask在识别物体时精度较低,速度慢.FCIS能够快速实现预测物体类别、包围框和生成掩膜的任务,但对重叠区域的预测结果不如Mask R⁃CNN.因此,本文选择Mask R⁃CNN作为分割网络,结果表明它在染色体分割任务中的效果很好.
在染色体分割问题上大规模应用深度学习算法的最大阻碍就是大规模标注数据的不足,因为在染色体中期图像这类显微镜拍摄的图像上进行像素级的标注非常费时费力.一种被广泛使用的数据标注方法是众包[1],但众包得到的标注数据的准确度很难保证,因为医学图像的标注需要操作者有丰富的医学知识和相关经验.随着生成式对抗网络(Generative Adversarial Networks,GAN)的出现,很多基于GAN网络的数据生成方法也被提出[11],然而这些基于GAN网络的方法只能合成语义分割的数据,不适用于实例分割.Dwidebi et al[12]提出一种通过自动融合前景和背景的方法来合成标注数据集,能快速合成大量数据.受此启发,本文提出一种自动生成像素级别标注的数据合成方法.实验证明,该方法有高度的真实性并能显著提高分割网络的准确度.
1.2 染色体分类
近年来,许多基于CNN的分类算法逐渐被提出.Sharma et al[1]第一次使用CNN进行染色体的分类.在他们的研究中,弯曲的染色体在分类之前会经过一次伸直预处理,该模型在1600张图片上进行训练,并在200张图片组成的测试集上取得86.7%的准确度.因为带标签数据的不足,Swati et al[16]提出一种基于孪生网络(Siamense Network)的分类方法,在稀少的训练集上取得了不错的效果,在209张图片的测试集上准确度达到84.6%.Wu et al[17]提出一种多分布生成式对抗网络(Multi⁃distribution⁃GAN,MD⁃GAN)来生成带标签数据,并使用一个预训练的CNN网络来进行分类,但准确度只有63.5%,远远低于临床要求.这些方法的一个共同问题就是带标签数据不足导致的过拟合、低准确度和低鲁棒性;而另一个不足是分类的准确度高度依赖于伸直算法的结果.基于多输入CNN,本文提出一种新的框架,能兼顾原始图片和伸直后图片的特征,提高了染色体分类的准确度和鲁棒性.
2 系统概述
传统的染色体核心分析流程如下:首先由医生使用光学显微镜拍摄染色体中期图像;然后,医生在辅助设备的帮助下对染色体中期图像进行筛选,挑选出可以进行核型分析的中期图像进行分割和分类;最后形成核型分析图片.整个过程需要医生花费大量的时间和精力人工进行核型分析工作,而且分割和分类的准确度受限于医生自身的工作熟练度以及状态,即精度不可控.为解决上述问题,提出一种基于卷积神经网络和几何优化的统计染色体核型分析方法,实现了染色体核型分析的自动化.
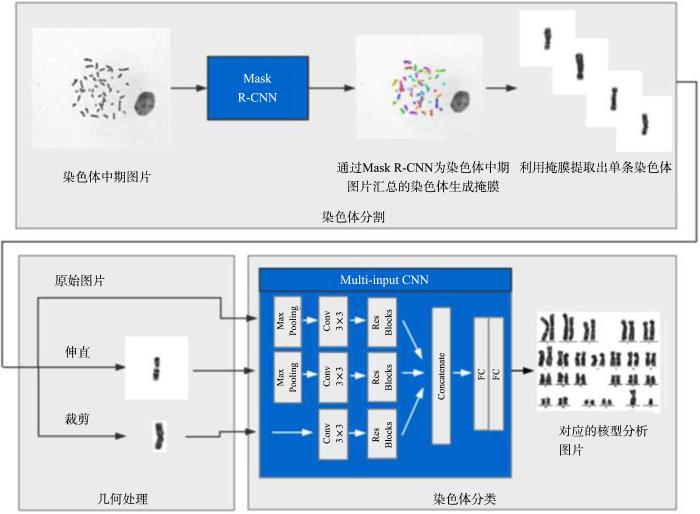
图2展示了系统的整个工作流程.第一阶段,将原始的染色体中期图像输入分割网络,再输出每个染色体掩膜,然后利用掩膜将染色体分别提取到子图像中.第二阶段,通过裁剪、旋转和矫直对每个子图像进行几何优化.最后将优化的子图像与原始提取的子图像一起输入分类网络.整个工作流程全自动,不需要人工干预.由于临床条件的高度复杂性和不确定性,在某些极端情况下该系统的准确性会有很大的差异,但我们的目标不是在核型分析中用系统完全替换医生,而是通过优化染色体核型分析来减轻医生的负担.
图1
图1
染色体中期图像(a)和对应的核型分析图片(b)
Fig.1
A metaphase chromosome image (a) and the corresponding karyotype image (b)
图2
图2
系统工作流程,包括三个阶段:染色体分割、几何优化和染色体分类
Fig.2
The pipeline of our system:chromosome segmentation,geometric optimization and chromosomes classification
3 数据收集和合成
本文使用的数据集分两部分:原始图片和合成图片.原始数据集由合作医院提供,并进行了
图3
图3
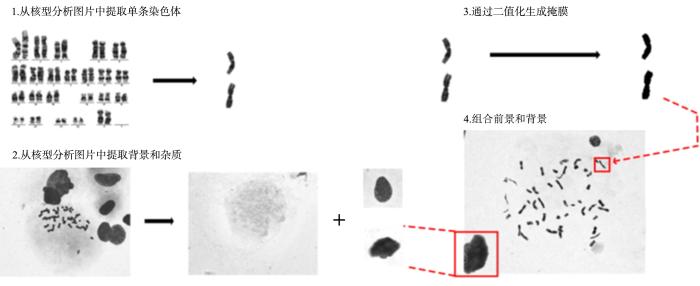
一种简单有效的染色体实例分割的像素级数据标注合成方法
Fig.3
A simple but effective method to synthesize pixel⁃level annotated images for chromosome instance segmentation
(1)收集染色体图片:从医院提供的核型分析图中直接提取染色体图片,共5000张128×128的单独染色体图片,每张包含一个染色体.
(2)收集杂质和背景:从染色体中期图像中收集50种不同的杂质图片和20个背景.
(3)前景掩膜生成:对每个染色体图片进行二值化和形态学处理,获得每个单独染色体的掩膜和最小轮廓.
(4)前景背景融合:随机选取48个染色体图片和2~6个杂质图片并粘贴到随机选择的背景上.为了防止不必要的覆盖,让染色体分布于图片的中心区域,杂质分布于四周.根据Dwibedi et al[12]的方法,使用多种融合方法能显著增加样本的真实性和可靠性,因此采用三种不同的图片融合方式:高斯模糊、动态模糊和线性滤波.
4 使用Mask R⁃CNN完成染色体分割
染色体分割任务中需要从染色体中期图像中分割出每一条染色体个体,但这些图片不仅包含染色体,还有各种杂质,染色体之间也经常接触和重叠,因此选用的分割网络需要完成以下任务:(1)目标检测,即能从杂乱的背景中检测出染色体区域;(2)像素级分割,即需要准确地分辨每个像素属于哪一条染色体,并对识别到的染色体生成对应的掩膜.本研究采用了Mask R⁃CNN.Mask R⁃CNN是一个通用的像素级目标分割框架,分三个部分:第一部分是共享的卷积层,用于特征图的获取,使用ResNet⁃101和FPN作为主干网络;第二部分是感兴趣区域(Region of Interest,ROI)提取网络(RPN),用于ROI的提取;通过ROI Align将每个感兴趣区域与特征图进行对齐输入第三部分完成掩膜的生成、分类和包围框生成的任务.Mask R⁃CNN的网络结构可以让其很好地完成目标检测、分类和像素级的目标分割任务.
5 基于中线的几何优化
为了解决这一问题,对现有的伸直算法进行了改进,具体如下.
5.1 中轴扩展
图4
图4
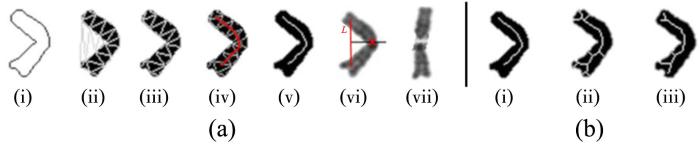
(a) (i)~(v)通过德劳内三角形获取染色体中线,
(vi)找到切割点,(vii)切割并旋转上下臂;(b) (i)为提取算法获取的中轴,(ii)和(iii)为scikit⁃image[18]的中轴提取算法
Fig.4
(a) (i)~(v) locate the medial axis using Delaunay triangulation,
(vi):find the cutting point, (vii) cut and rotate the upper and lower parts, (b) (i)our medial axis locating algorithm,(ii) and (iii) medial axis locating algorithms provided by scikit⁃image[18]
5.2 切割点寻找
5.3 旋 转
找到弯曲切割点
5.4 像素填充
因为想在处理后的图像中最大化水平条带特征,算法使用了一种新的插值方法.对图像I中第m行的每一个待填充像素,有:
其中,
6 使用多输入CNN继续染色体分类
受He et al[21]提出的残差网络的启发,使用ResNet⁃50作为特征提取网络.原始图像和伸直后的图片首先经过一个2×2的最大池化层,然后和裁剪后的图片一起输入残差网络,这样能使特征提取最大化.在CNN网络收敛之后,全局特征、局部特征和伸直后的特征被提取并送入分类器.为了最大化地利用这些特征,使用一个concatenate层进行特征融合,并使用一个由两层FC层组成的分类器来学习这些融合特征和具体类别之间的对应关系.
为了使这个模型效果最好,先训练三个单独的CNN模块直到收敛;然后把三个网络拼接在一起,固定CNN的参数,只训练顶层的分类器;最后把整个模型解锁,并在一个很低的学习率(0.0005)下进行微调.
7 结果和讨论
7.1 分割结果
通过用多种比率混合(混合时保持343张手工标注数据不变)的真实数据和手工标注数据设置六个训练数据集和两个测试数据集,单独地在每个训练集上训练六个模型.我们的模型以Abdulla[22]的开源代码为基础,在训练过程中针对染色体分割任务调整了部分超参数:RPN的一系列候选区域大小设置为
分割的实验结果如表1所示,黑体字表示上述测试集测试下的最优结果.
表1 分割网络在不同数据集上训练的模型在测试集上的测试结果(%)
Table 1
| 数据集 | 测试集1( | 测试集2( | 测试集1( | 测试集2( |
|---|---|---|---|---|
| 343张手工标注图片 | 52.059 | 44.488 | 90.590 | 90.010 |
| 343张合成图片 | 47.563 | 31.805 | 88.194 | 76.967 |
| 1000张合成图片 | 53.476 | 35.450 | 91.657 | 83.855 |
| 手工标注和合成图片比例为1∶1 | 57.827 | 41.882 | 94.030 | 91.931 |
| 手工标注和合成图片比例为3∶7 | 57.841 | 43.248 | 94.563 | 91.673 |
| 手工标注和合成图片比例为1∶4 | 59.998 | 44.794 | 95.644 | 91.662 |
设置两个测试集:测试集1由100张手工标注数据组成,测试集2由100张手工标注数据和100张合成数据混合组成.
图5
7.2 分类结果
使用48万张图片作为训练集,验证集和测试集各9万张.所有图片都经过伸直和裁剪处理,并将原始图片和裁剪伸直后的图片同时输入网络.使用
表2展示了单个CNN和综合CNN(Global⁃Net,Local⁃Net,straight⁃net和组合模型)的实验结果.可以看到,多输入CNN的准确率比任何一个单一CNN都高,因为多输入CNN能提取更多特征,因此鲁棒性较高,这意味着多输入模型可以通过对组合特征的预测来提取更多的特征,减少分类误差.
表2 单个CNN和组合CNN的准确率比较
Table 2
| 方法 | 准确率 |
|---|---|
Global⁃only Local⁃only Straighten⁃only Global & Local All⁃combined | 0.9445 0.9200 0.8200 0.9455 0.9570 |
表3 我们的方法与一些常见模型的准确率比较
Table 3
| 方法 | 准确率 |
|---|---|
| AlexNet | 0.8975 |
| VGG⁃16 | 0.9023 |
DenseNet ResNet⁃50 Sharma et al[1] Our model | 0.9414 0.9445 0.9012 0.9570 |
表4显示了每一类染色体的分类情况,表中黑体字表示三种评价标准下每类染色体的最优结果.可以看出,我们的模型在所有分类上都有比较优秀的表现:在第一类染色体上的F1分数甚至达到了0.9901;即使是表现最差的Y染色体,F1分数也仍然达到0.92.
表4 分类模型在每一类上的表现
Table 4
| 染色体类别 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 准确率 | 0.9941 | 0.9737 | 0.9862 | 0.9370 | 0.9611 | 0.9677 | 0.9615 | 0.9191 | 0.9251 | 0.9401 | 0.9715 |
| 召回率 | 0.9861 | 0.9927 | 0.9887 | 0.9554 | 0.9295 | 0.9841 | 0.9619 | 0.9337 | 0.9246 | 0.9446 | 0.9729 |
| F1分数 | 0.9901 | 0.9831 | 0.9875 | 0.9461 | 0.9450 | 0.9758 | 0.9615 | 0.9267 | 0.9248 | 0.9423 | 0.9426 |
| 染色体类别 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 | 22 |
| 准确率 | 0.9681 | 0.9681 | 0.9322 | 0.9490 | 0.9639 | 0.9693 | 0.9643 | 0.9426 | 0.9644 | 0.9425 | 0.9291 |
| 召回率 | 0.9678 | 0.9813 | 0.9397 | 0.9406 | 0.9639 | 0.9726 | 0.9469 | 0.9571 | 0.9571 | 0.9560 | 0.9301 |
| F1分数 | 0.9644 | 0.9747 | 0.9360 | 0.9448 | 0.9639 | 0.9711 | 0.9556 | 0.9316 | 0.9608 | 0.9492 | 0.9296 |
| 染色体类别 | X | Y | AVG | ||||||||
| 准确率 | 0.9493 | 0.9200 | 0.9567 | ||||||||
| 召回率 | 0.9407 | 0.9327 | 0.9552 | ||||||||
| F1分数 | 0.9450 | 0.9263 | 0.9552 |
8 结 论
提出一种利用深度学习和几何优化的染色体核型自动分析方法,首次使用深度学习方法进行染色体分割.提出一种简单高效的染色体分割标注数据自动生成方法,对分割数据集进行扩充,并验证了合成数据的可用性,为染色体分割领域的进一步研究奠定了基础.使用Mask R⁃CNN来实现染色体分割的自动化,取得了较好的效果.对于高度弯曲的染色体,提出一种比现有方法的通用性更好的优化算法.在此优化算法的基础上构建一个多输入CNN对染色体进行分类,准确率为95.6%,远高于现有的方法.本研究为染色体自动核型分析系统的实现提供了一种思路.
参考文献
Crowdsourcing for chromosome segmentation and deep classification
∥
Mask R⁃CNN
∥
Conference on Computer Vision
Current methods in medical image segmentation
Trainable grey⁃level models for disentangling overlapping chromosomes
Topology aware fully convolutional networks for histology gland segmen⁃tation
∥
Fully convolutional networks for semantic segmentation
∥
U⁃net:convolutional networks for biomedical image segmen⁃tation
∥
Fully convolutional instance⁃aware semantic segmentation
∥
Learning to refine object segments
∥
Generative adversarial learning for reducing manual annotation in semantic segmentation on large scale miscroscopy images:automated vessel segmentation in retinal fundus image as test case
∥
paste and learn:surprisingly easy synthesis for instance detection
∥
Outliers in statistical pattern recognition and an application to automatic chromosome classification
Medial axis transform⁃based features and a neural network for human chromosome classification
Automatic chromosome classification using support vector machines
.
Siamese networks for chromosome classification
∥
End⁃to⁃end chromosome karyotyping with data augmentation using gan
∥
Scikit⁃image:image processing in python
A new approach to automatic classification of the curved chromosomes
∥
Multi⁃scale convolutional neural networks for lung nodule classification
∥
Deep residual learning for image recognition
∥
Mask R⁃CNN for object detection and instance segmentation on keras and tensorflow