不同的分子机制决定了不同的转录动力学,如转录爆发的频率调控和幅度调控等,导致转录产物丰度和持续时间的分布更加多样化.这更需要准确的模型给出定量化的解释,给出新的可供实验检验的理论预言.本文综述近年来的转录爆发模型,分析各模型的优缺点.
1 转录爆发与不应期
先简介基因的转录爆发现象.
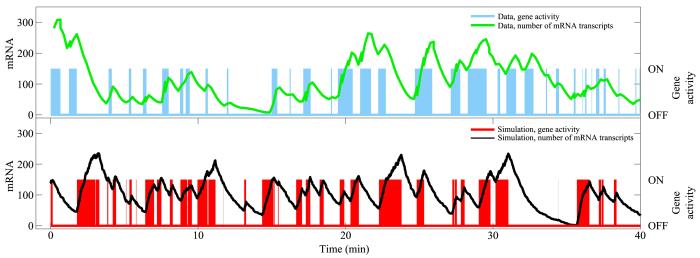
转录过程曾被认为是一个平稳的过程,即单位时间内产生的mRNA数量是在一个平均值附近的小幅扰动,因此转录速率是常数,mRNA的产生是个泊松过程.传统的实验是针对细胞群体的测量,只保留了转录中mRNA的平均信息,丢失了单细胞的动态信息.随着单细胞技术的发展[18,19],尤其是单分子荧光原位杂交(Single⁃mo⁃lecule Fluorescence in situ Hybridization,SFSH)技术的出现,新生成的mRNA可以被追踪.人们发现,除了管家型基因[20],很多基因的转录其实是间歇性的爆发过程,而非传统认为的泊松过程[11].基因开始转录时会从沉默态(无转录活性)进入一个相对短暂的激活态,快速并大量地生成mRNA,再重新回到沉默态;该过程重复出现,直到转录信号消失或转录过程被阻遏.经过剪切加工,初生的mRNA变为成熟的mRNA、出核,而细胞核内的pre⁃mRNA也会被降解,这些都导致核内mRNA的减少.从低等的原核生物到高等的哺乳动物[9,10,11,12],这一现象广泛存在,说明转录爆发是基因表达的一种基本模式.图1表达了单细胞中的转录爆发过程(基因Prl2c2的实验数据和模拟结果),整个过程只有一个稳定的转录信号,没有考虑对mRNA降解速率的调控.图中蓝线和绿线分别是实验得到的Prl2c2转录活性和mRNA数量随时间的演化曲线[12];红线和黑线是基于两态模型和Gillespie算法[21]模拟得到的(
图 1
其中,
考虑最简单的情况:只有一个
同理可以得到
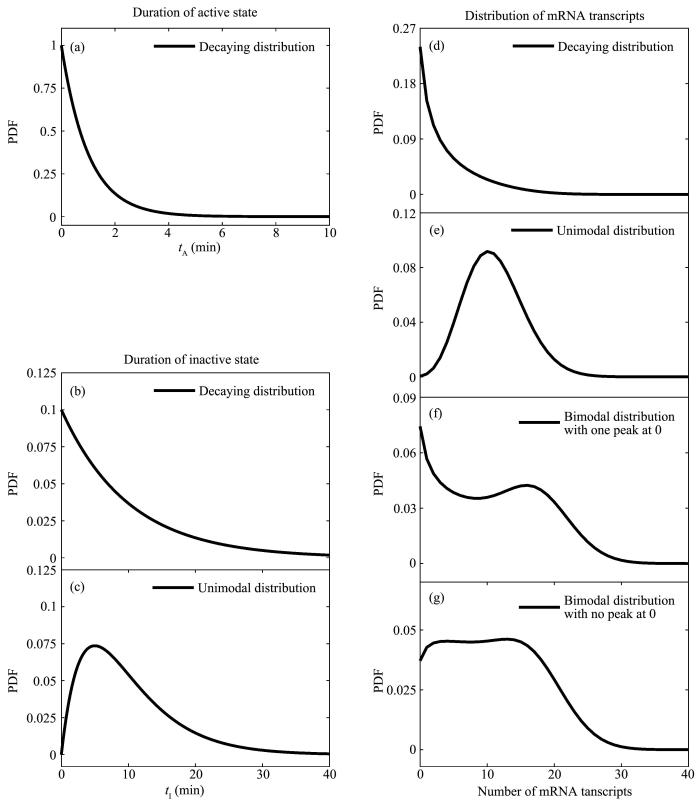
图2
图2
转录爆发的典型特征
Fig.2
Typical features of transcriptional bursting
Schematically shown are the distributions of duration of active and silent gene states (Fig.2a~2c) and of the number of mRNA transcripts (Fig.2d~2g) under different conditions. PDF refers to probability density function.
模型得到的(
2 转录模型
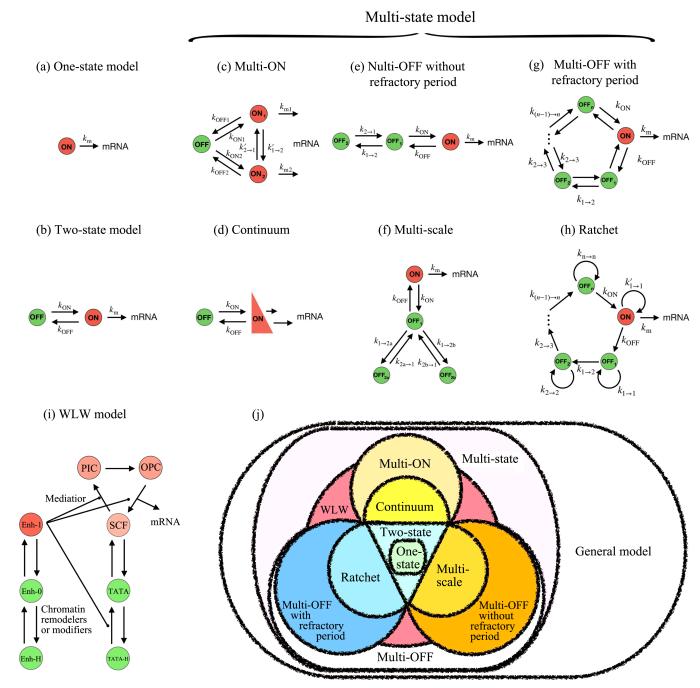
图3
图3
转录模型
Fig.3
Modeling of transcription
Shown are the one⁃state (a),two⁃state (b),multi⁃ON (c),continuum (d),multi⁃OFF without necessary refractory period (e),multi⁃scale (f),multi⁃OFF with necessary refractory period (g),ratchet (h) and WLW (i) models. (j) shows model compatibility.
2.1 单态模型
在单态模型(图3a)中,转录是泊松过程[8].转录速率是常数,其数值依赖于调控信号的强度,常用希尔函数来刻画[29].这一简化模型只保留了mRNA在细胞群体水平的平均信息,包含两个参数:转录速率常数km和降解速率常数δ.当研究mRNA或者蛋白质水平且它们的相对噪声较小(CV较小)时,可以采用单态模型.比如,当蛋白质的降解速率较慢,蛋白质丰度的CV较小,就常采用这一模型,甚至将转录和翻译过程合并在一起考虑.该模型简单、参数少,适合用于研究复杂的细胞信号转导网络;缺点是太过简单,失去了基因转录的绝大部分信息.2.2 两态模型两态模型(又称ON⁃OFF模型,图3b)向下兼容单态模型,向上又被多态模型所兼容,是当前广为使用的唯象模型[14,30].两态指基因启动子的两个状态:激活态(ON)和沉默态(OFF).模型包含四个参数:从沉默态到激活态的转换速率常数
当mRNA数目满足泊松分布、调控信号影响主要限速步骤(持续时间呈指数分布),两态模型可以很好地描述mNRA数量和基因活性的随时演化等,也适用于描述单态模型适用的情形,但计算量更大.果蝇间隙(gap)基因中由统一的转录动力学机制导致的不同空间图样就可以用两态模型很好地描述[26].当mRNA数目呈多峰分布、转录受多种信号调控或者调控信号作用于非限速步骤(持续时间偏离指数分布)时,两态模型就不再适用.两态模型是唯象模型,其参数包含了整体的均值信息,往往刻画的是关键的限速步骤,但忽略了许多细节.
2.3 多态模型
多态(multi⁃state)模型是在两态模型的基础上发展起来的.它包含多个启动子活性状态,可描述持续时间偏离指数分布、呈单峰或双峰分布的情形,以及mRNA数目呈双峰分布的情形.简单来说,就是激活态或沉默态是有记忆的.mRNA的产生涉及多个反应步骤,单分子事件之间存在关联(记忆).这涉及阻遏物、转录因子和中介物的复杂调控过程以及染色质重构或组蛋白修饰等,其中的许多分子机制还远不清楚.
从两态模型出发,可分别将ON态和OFF态细分,得到多ON态[15,32]和多OFF态[16,22,23,32]两大类唯象模型(图3c至图3h).各个状态受限速步骤的影响,但并不一一对应(可能是多个步骤的合并).激活态的时长呈指数或近指数分布时,如果不考虑激活态中子状态的差异(或差异不大),多ON态可以合并为单ON态,所以一般在考虑RNA聚合酶状态时才使用多ON态模型.对许多基因,如与催乳素[33]基因相关的启动子表现出很强的记忆性,不应期时长呈单峰分布,多OFF态模型更常见.模型的参数包括:从沉默态到激活态的转换速率常数
多ON(multi⁃ON)[15,17]模型(图3c)考虑的是存在多个ON态的情形.影响ON态数量的因素很多,如转录因子的空间分布、TAD(Topologically Assocaited Domain)和RNA聚合酶的相分离等.当ON态是由大量受转录因子或表观遗传标记的特异性结合所定义的子状态组成,每个子状态具有不同的启动速率,并且在时间上紧密相邻,或者ON态中启动子受RNA聚合酶局部时变浓度影响、导致大量准连续的启动速率时,可以用连续性(continuum)模型[34](图3d)来刻画转录过程.连续性模型主要考虑启始转录速率的波动,涉及多种因素,如RNA聚合酶的相分离、转录因子的空间分布等.两个连续启动事件间隔几秒到十多秒不等;这些间隔不服从单指数分布,而是服从大量的指数分布,其期望值是准连续分布的.因此,相应的起爆速度几乎跨越连续的区间.从本质上来讲,不管是考虑ON态中的子状态还是考虑RNA聚合酶的时空分布,连续性模型都应该归为多ON态模型,但由于ON态的持续时间变化不大,连续性模型又可以看成是参数可变的两态模型.连续性模型适合于研究由时空特异性导致、拥有不同分子结合速率的转录过程.
根据是否存在基因启动子重新激活前必须经历的不应期,多OFF态模型又分成无不应期[32](图3e)和含不应期[16,22,23](图3g)的两类模型.无不应期模型有更多可能的启动路径,很容易延伸发展为多尺度(multi⁃scale)模型[35](图3f).多尺度模型考虑的是基因在再次激活前,存在多条路径且耗费的时间长短不同.举例来说,考虑结合在启动子上的脚手架结构(Scaffold Complex,SCF)的不完全拆解和完全拆解,就可以导致不应期存在多个时间尺度.含不应期的multi⁃OFF模型存在很强的不可逆性;如果沉默态足够多,模型还会呈现出很强的周期性.考虑到转录过程存在可替代的路径(分岔)以及微弱的可逆反应,模型可进一步发展为棘轮模型[36,37,38](图3e).在OFF态除了沿着主要方向进行的反应,其余分岔和可逆反应都简化为反应常数为
相比两态模型,多态模型对mRNA分布的刻画更准确,更适用于研究信息的传递,能够更好地解释许多实验现象.Wang et al[39]提出的模型(简写为WLW模型)(图3i)是从基础的转录机制来解释转录爆发,揭示了转录的生物化学机制对转录动力学的影响.它将转录中启动子的变化分成三部分:mRNA生成、脚手架结构的装配与拆解、组蛋白的装配和修饰,并通过激活子的结合与解离来控制这三个部分.WLW模型考虑的是含TATA框的基因转录过程,其中最重要的就是增强子(enhancer)和TATA框上的状态变化.增强子可以是被组蛋白占据的状态(Enh⁃H)、裸露的状态(Enh)或者被转录因子占据的状态(Enh⁃1).结合在增强子上的激活子通过使脚手架结构中的媒介子(Mediator)异构化[40,41]来调控起始转录速率,将信息传递给Pol II.这意味着ON态的持续时间和转录速率是耦合在一起的,难以像其他的唯象模型将两者分离.当激活子结合在增强子上时,通过招募染色质重构酶[4,5]和修饰酶[6,7],使得占据TATA框的组蛋白很容易被清除,为脚手架结构的构建做好准备.转录因子是否占据增强子以及转录因子的种类则会影响TATA框上状态转变的速率,而转录因子的浓度越高,增强子被其占据的频率越高,导致转录速率、激活态和沉默态时长是转录因子浓度的函数.与增强子相对的是沉默子,能够产生相反的效果.如果没有TATA框,WLW模型的结构也可以用,只不过反应过程有所不同:没有TATA框,不再是TFIID中的TATA框结合蛋白(TATA⁃box binding protein,TBP)与TATA框结合,而是TFIID中其他亚基结合到启动子上.其实,可将WLW模型中TATA框的概念延申为代指或标识核心启动子区域.
总结一下,单态模型是最简单的模型,主要刻画mRNA的均值.它能发展为两态模型,为两态模型兼容(图3j).在合适参数下,两态模型可以退化为单态模型.两态模型能刻画转录爆发大小和频率的均值,以及部分呈指数分布的基因激活态和沉默态持续时间.研究mRNA的分布及其对信号的响应时,常常要运用多态模型.多态模型分为多ON态和多OFF态模型,它们兼容单态和两态模型.当研究具体的转录机制时,多ON态模型可发展为连续性模型.而多OFF态模型根据是否存在必须经历的不应期,分成两类;考虑到具体的分子机制,两者又可以分别发展为棘轮模型和多尺度模型.连续性模型侧重于刻画转录因子或聚合酶的局部时空变化对转录的影响,可用于研究相分离对转录的影响.棘轮模型主要研究不可逆反应和路径分岔的影响,适合于研究转录中的能耗.多尺度模型侧重于脚手架结构对转录的影响,适合研究温度、启动子序列、转录因子等对转录的影响.WLW模型考虑作用于增强子区域的转录因子,突出的是转录因子自身生物功能对转录过程(对增强子、TATA框和Pol II的状态)的影响.只要把不断招募Pol II的过程合并为ON态,再把增强子、TATA框的状态向量与OFF态对应,WLW模型就可以转化为考虑基因状态的模型,归并到多态模型里,但是激活态时长与转录速率耦合在一起.事实上,WLW模型只要多考虑一些转录因子、染色质的重构与修饰反应,就能再现多态模型中的复杂动力学,兼容连续性模型、多尺度模型和棘轮模型.当然,受限于内在的转录机制,WLW模型不能模拟一些细菌基因的动力学.
此外,有外部信号(如转录因子,Transcription factor,TF)调控转录时,实际上有两组参数,分别对应有TF结合和没有TF结合的情形.假设TF的结合可以提高转录水平,那么在没有TF结合时,转录事件发生的频率很低,可以视为泊松过程.在保证模拟结果大致不变的情况下,对单态模型、两态模型和多态模型而言,没有TF结合的一组参数常常可以简化为一个参数(
上述的单态、两态、多态模型,只要适当变化一下,与Zhang and Zhou[15]提出的一般模型是相容的(图3i),且参数个数和计算量不断增加.针对mRNA均值、单细胞mRNA的动力学和分布,用单态模型、两态模型以及多ON和多OFF模型来预测和解释现象就够了.当研究的问题与转录机制、能耗、具体的信号调控、信号转导过程有关时,就要运用连续性模型、棘轮模型、多尺度模型和WLW模型了.很多情况下,这些模型模拟的结果与多ON或多OFF模型的结果相差不大(在实验误差内),但是添加转录的分子机制势必会增加复杂性和计算量,所以往往只有在研究具体分子机制或者信息传输时,才采用这些模型.在保证模型能够表达研究所需转录信息的情况下,则一般选择更加简单的模型.当出现新的现象、原有模型无法解释时,先尝试能够兼容原模型的已有模型,失败后再尝试其他的现有模型.如果都失败,再基于两态模型,根据激活态和沉默态时间的分布、转录爆发大小以及mRNA数量分布随调控因子的变化,发展新模型.
3 数据分析与数值模拟
确定模型后便可根据实验数据确定模型参数、开展数值模拟、解释实验现象或预测新结果.
3.1 数据分析
实验给出的数据多种多样,可以是蛋白质或mRNA信息、核内或核外信息、群体或单细胞信息.这里主要介绍如何对单细胞中ON态时长、OFF态时长、mRNA数量等进行分析.如果这三组数据只是在稳态下测得的,那就已经丢失了部分转录信息,无法还原所有转录细节.用这三组数据可分别得到激活态和沉默态持续时间、mRNA数量的平均值,即
不妨以研究转录因子浓度对转录的影响为例来说明.首先,根据转录爆发大小的变化,判断由激活态进入沉默态的概率
3.2 数值模拟
对模型构建后的数值模拟,主要通过化学主方程来计算分布信息,用Gillespie算法[21]来模拟基因状态和mRNA的时变轨迹.所有模型适当变化后都可以转变为态模型.假设模型中共有N个状态,
通过上式,便能模拟概率分布随时间的变化.如果要得到单细胞中mRNA的时序图,就要使用Gillespie算法.
4 总 结
表1 转录模型的适用性
Table 1
| 模型 | 单态模型 ( | 两态模型 ( | 多态模型(图3c至图3i) | ||
|---|---|---|---|---|---|
多ON模型 (含连续性模型) | 多OFF模型 (含棘轮模型和多尺度模型) | WLW模型 | |||
| 激活态的持续时间分布 | / | 主要时 | 主要时 | ||
| 沉默态的持续时间分布 | / | ||||
| mRNA数量分布 | |||||
保 留 信 息 | 细胞群体水平的平均mRNA信息 | ||||
| mRNA的噪声强度、爆发频率和大小、激活态和沉默态的平均持续时间 | |||||
| mRNA分布性质 | |||||
| 激活态持续时间分布 | 沉默态持续时间分布 | 激活态和沉默态的 持续时间分布 | |||
| 精确度 | 低 | 中等 | 较高 | 较高 | 高 |
| 参数 | 很少,可由实验测得 | 少,可由实验测得 | 中等,部分可由实验测得,部分需要假设且要与实验相符 | 中等,部分可由实验测得,部分需要假设且要与实验相符 | 多,主要步骤可由实验测得,部分未明确的机制需要假设 |
| 计算量 | 很小 | 小 | 中等 | 中等 | 大 |
| 适用情况 | mRNA或蛋白质的CV较小,不考虑单细胞中mRNA的时变 | 研究单细胞转录产物数量变化,基因状态切换以及转录调控等 | mRNA数量呈多峰分布、激活态时长呈单峰分布,或者研究转录调控和信号传导时采用多ON态模型 。在研究相分离对转录的影响时可用连续性模型,侧重于转录因子或者聚合酶的局部时空变化对转录的影响 | 研究周期性和能耗时,可采用棘轮模型。研究温度、启动子序列、转录因子等对转录的影响时用多尺度,侧重于脚手架结构对转录的影响 | 研究转录因子自身生物功能对转录过程的影响 |
构建转录模型,主要是为了能够定量解释实验现象,提供可供实验检验的理论预言.当研究对象是细胞系或者受精卵时,考虑细胞分裂[42]、基因复制[43]和体积变化[44]时,可根据研究的需要选择不同的模型.构建转录模型受到研究对象所需模型层次的影响.转录调控模式、包含转录的信号传导等问题是当前的研究热点,往往需要精确的转录模型,更多地与转录机制相结合.随着更多蛋白质标签和有机染料的开发利用[45,46,47],会有更多的单细胞数据出现,更多的蛋白质工作机理将被揭示,促进对转录机制的理解.构建转录模型,解释和预测转录爆发动力学仍将是一个重要的研究方向.构建更详细的转录模型十分依赖实验数据,依赖于科学技术.利用分子模拟、力化学技术、单分子荧光原位杂交技术(smFISH)、荧光成像(Fluorescence Imaging)以及二项分(Binomial Partitioning) [48],能够得到单分子作用形式和单细胞中特定基因的转录轨迹.困难在于转录过程涉及太多的反应、分子作用机制多样、各种信号干扰,限制逆推模型结构.因此,设计能够精准调控单步反应的实验和发展有效处理含噪声数据的数学方法是必须的.此外,研究转录对于外界信号(随时空变化)的响应,也是一个重要的方向.它将阐明外界信息是如何通过存在不确定性的转录过程传递下去的,证明转录精巧性的一面.
参考文献
Genome⁃wide dynamics of Pol II elongation and its interplay with promoter proximal pausing,chromatin,and exons
Regulation of RNA polymerase II activation by histone acetylation in single living cells
Transcription factors modulate c⁃Fos transcriptional bursts
Dynamic regulation of transcriptional states by chromatin and transcription factors
Linking stochastic fluctuations in chromatin structure and gene expression
Modulation of transcriptional burst frequency by histone acetylation
Methylation of H3K4 is required for inheritance of active transcriptional states
Stochastic models of transcription:from single molecules to single cells
Transcriptional pulsing of a developmental gene
Real⁃time kinetics of gene activity in individual bacteria
Stochastic mRNA synthesis in mammalian cells
Mammalian genes are transcribed with widely different bursting kinetics
Gene transcription in bursting:a unified mode for realizing accuracy and stochasticity
Induction mechanism of a single gene molecule:Stochastic or deterministic? BioEssays
,
Promoter⁃mediated tran⁃scriptional dynamics
Analytical distribution and tunability of noise in a model of promoter progress
Analytical results for a multistate gene model
Localization of ASH1 mRNA particles in living yeast
Visualization of single RNA transcripts in situ
Single⁃RNA counting reveals alternative modes of gene expression in yeast
Exact stochastic simulation of coupled chemical reactions
Effects of molecular memory and bursting on fluctuations in gene expression
Structure of silent transcription intervals and noise characteristics of mammalian genes
Measurement of gene regulation in individual cells reveals rapid switching between promoter states
Estrogen-dependent control and cell⁃to⁃cell varia⁃bility of transcriptional bursting
Diverse spatial expression patterns emerge from unified kinetics of transcriptional bursting
Bimodal gene expression in noncooperative regulatory systems
Noise can induce bimodality in positive transcriptional feedback loops without bistability
Nature,nurture,or chance:stochastic gene expression and its consequences
Markovian modeling of gene⁃product synthesis
Transcriptional burst frequency and burst size are equally modulated across the human genome
Intrinsic dynamics of a human gene reveal the basis of expression heterogeneity
Dynamic analysis of stochastic transcription cycles
A continuum model of transcriptional bursting
A single⁃molecule view of transcription reveals convoys of RNA polymerases and multi⁃scale bursting
On the way of revealing coactivator complexes cross⁃talk during transcriptional activation
Sequential recruitment and combinatorial assembling of multiprotein complexes in transcriptional activation
Reconciling the concurrent fast and slow cycling of proteins on gene promoters
Dynamic mechanism for the transcription apparatus orchestrating reliable responses to activators
Mediator and the mechanism of transcriptional activation
Dynamic regulation of pol II transcription by the mammalian Mediator complex
Random partitioning of molecules at cell division
Effects of DNA replication on mRNA noise
Single mammalian cells compensate for differences in cellular volume and DNA copy number through independent global transcriptional mechanisms
Single⁃molecule dynamics of enhanceosome assembly in embryonic stem cells
Single⁃molecule analysis of steroid receptor and cofactor action in living cells
A general method to improve fluorophores for live⁃cell and single-molecule microscopy
Figure 1 theory meets figure 2 experiments in the study of gene expression